© Olivier GASCUEL / Matthieu JUNG / Martine PEETERS / C3BI / IRD / CNRS Images
Référence
20170075_0001
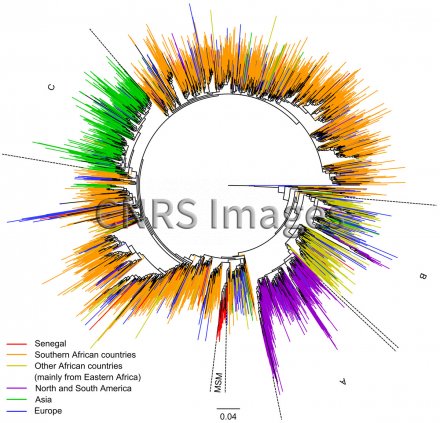
Arbre phylogénétique décrivant la propagation du sous-type C du VIH-1
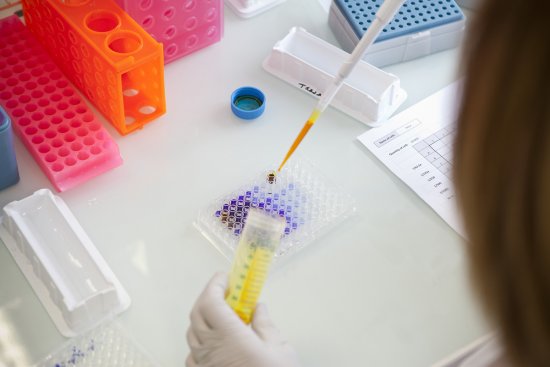
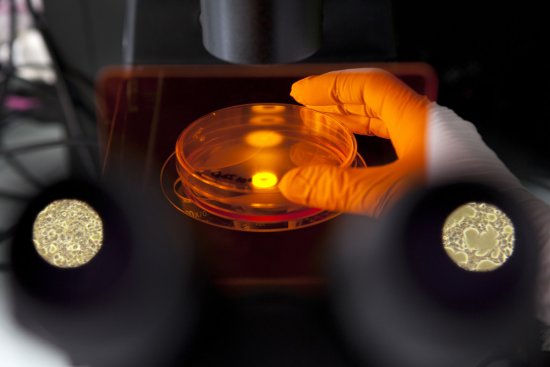
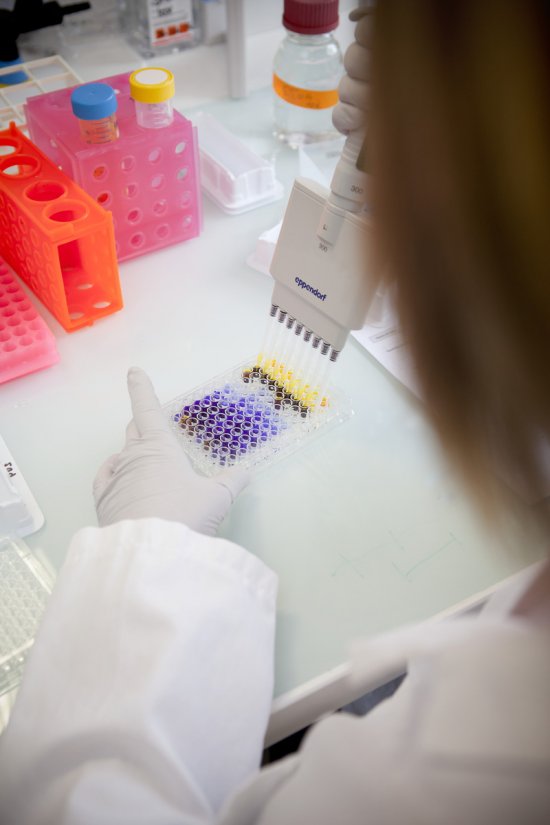
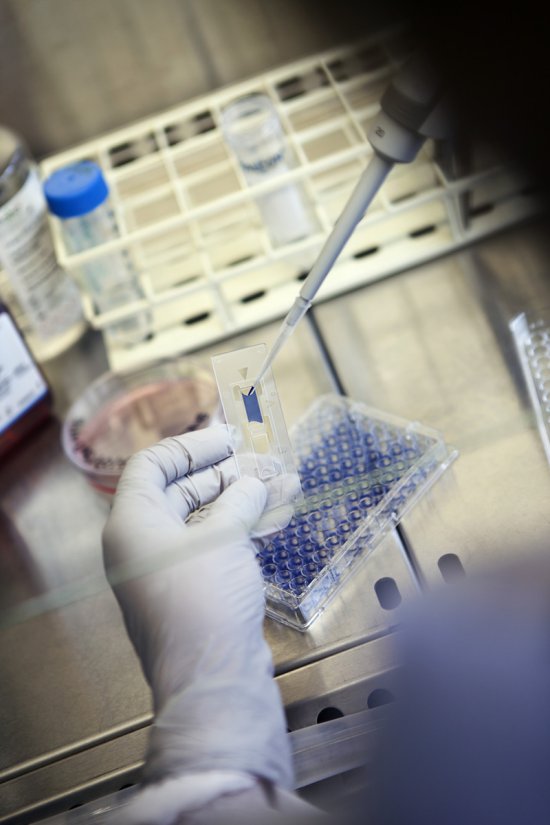
Arbre phylogénétique décrivant la propagation du sous-type C du VIH-1, généré via le logiciel PhyML à partir de plus de 3 000 séquences du virus provenant du monde entier. L’arbre décrit globalement l’évolution des séquences, le système de coloration est utilisé pour corréler cette évolution à la géographie et aux facteurs de risque. Chaque branche (ligne colorée) représente l’évolution d’une séquence virale. La longueur des branches varie en fonction du nombre estimé de mutations. Les jonctions entre les branches correspondent à des transmissions inter-patients ou à des diversifications intra-patients. L’échelle 0,04 correspond à 4% de mutations par nucléotide. Le taux moyen de mutations étant d’environ 0,003 mutations par nucléotide et par an, cette échelle représente ainsi un peu plus de 10 ans d’évolution. Les lettres A, B, C sont des sous-groupes du sous-type C réunissant des souches qui correspondent à des isolats géographiquement liés. C’est le cas pour la souche A, principalement identifiée en Amérique et pour la souche C, surtout présente en Asie. Cette homogénéité traduit une introduction récente et parfois unique, par exemple en Asie, au contraire de l’Afrique et de l’Europe. La portion rouge, MSM ou HSH (Hommes ayant des rapports sexuels avec des hommes), montre qu’au Sénégal l’introduction du sous-type C a elle aussi été unique dans cette population spécifique. L’étude des différentes introductions du VIH-1 à l’échelle des pays et des continents et au sein des groupes de risque, permet une meilleure compréhension des mécanismes épidémiques et un meilleur contrôle de la propagation du virus.
L’utilisation des médias visibles sur la Plateforme CNRS Images peut être accordée sur demande. Toute reproduction ou représentation est interdite sans l'autorisation préalable de CNRS Images (sauf pour les ressources sous licence Creative Commons).
Aucune modification d'une image ne peut être effectuée sans l'accord préalable de CNRS Images.
Aucune utilisation à des fins publicitaires ou diffusion à un tiers d'une image ne peut être effectuée sans l'accord préalable de CNRS Images.
Pour plus de précisions consulter Nos conditions générales